Het Resultaat
Deze pagina zal in het teken staan van de resultaten. De resultaten staan gerangschikt op het moment van uitvoeren. Hierbij is het handig om te weten dat de proefpersonen een letter van A tot en met H hebben gekregen. In de tabel hiernaast is te zien tot welke interventiegroep deze personen horen. Persoon E maakte hierbij geen deel uit van de interventie maar hier zijn wel resultaten van te vinden.
| Persoon | Interventie groep |
|---|---|
| A | Yakult |
| B | Yakult |
| C | Yakult |
| D | Listerine Zero |
| E | - |
| F | Listerine Zero |
| G | Controle |
| H | Yakult |
De voedingsbodems
Onze resultaten beginnen bij de voedingsbodems. We hebben hier drie verschillende soorten van de bloedagar waar alles op groeit, de MS agar en de MRS agar. Helaas is er op de MS agar niks gegroeid omdat deze te lang in de koelkast heeft gestaan en daarom is deze plaat zijn werking is verloren. De bloedagar is gebruikt voor de telling van de kolonie vormende eenheden, deze staat hieronder. Maar hier zijn ook drie verschillende kolonies vanaf gehaald. 3.1 De witte kolonie, 3.2 de groene kolonie en 3.3 de grijze kolonie met de zwarte stip in het midden. En dan is er ook nog de MRS agar gebruikt. Deze had wel groei en met de kolonies die hierop zaten hebben we ook het moleculaire gedeelte van ons onderzoek gedaan. Hiermee hebben we dus PCR's uitgevoerd, op gel gebracht en laten sequensen.
De Tellingen
Het aantal KVE (kolonie vormende eenheden) zijn geteld op 2 momenten voor de interventie en na de inverntie. De totale aantallen zijn met elkaar vergeleken, met een bijbehorende procentuele toe of afname. Deze zijn in de tabel hiernaast te zien. Zo kan er dus worden bekeken of er een (te verwachte) afname heeft plaatsgevonden bij de Yakult en de Listerinegebruikers. Veelal is er een afname waargenomen bij de Yakult en Listerine gebruikers.
De conclusie is dat er bij bijna iedereen een daling heeft plaatsgevonden. Het maakte geen verschil in welke groep deze personen zich bevonden.
| Persoon | KVE/ml Voor | KVE/ml Na | Toe- of afname |
|---|---|---|---|
| A | 290.000 | 7.300 | -97.48% |
| B | 480.000 | 5.000 | -99.00% |
| C | 37.000 | 410 | -98.90% |
| D | 29.000 | 90.000 | +210.34% |
| F | 610.000 | 700 | -99.90% |
| G | 1.500.000 | 4.800 | -99.70% |
| H | 240.000 | 3.900 | -98.40% |
De Gramkleuring
De gramkleuring is na de groei van de bacteriën op de plaat uitgevoerd, om te kijken of de op het oog waar te nemen bacterie wel degelijk de bacterie is die we denken dat het is. Hierin verwachten we dat S. mutans, de witte kolonie zal zijn en onder de microscoop een positieve kok zal tonen. Dit is onder de microscoop dan 'een rondje' en paars van kleur. Verder wordt er verwacht dat L. casei de groene kolonie zal zijn en onder de microscoop een positieve staaf zal tonen. De gramkleuring is voor P. gingivalis niet uitgevoerd omdat de kolonie ervan een duidelijk zwart puntje is.
Bij alle witte kolonies was er een positieve kok waargenomen, maar bij een groene kolonie is over het algemeen een positieve kok te zien waar we een staaf verwachtten. Dit kan komen doordat er andere bacteriën in de mond zitten die groene kolonies vormen.
In de onderste tabel staan de uitslagen van de stammen waar we naar opzoek zijn. Hieruit blijkt dat we mogelijk in de witte kolonie de S. mutans kunnen vinden.
| Persoon | Witte kolonie | Groene kolonie |
|---|---|---|
| A | Positieve kok | Negatieve kok |
| B | Positieve kok | Positieve kok |
| C | Positieve kok | Positieve staaf |
| D | Positieve kok | Positieve Staaf |
| E | Positieve kok | Negatieve kok |
| F | Positieve kok | Positieve kok |
| G | Positieve kok | Positieve kok |
| H | Positieve kok | Positieve kok |
| Controle stam | Uitslag |
|---|---|
| P. gingivalis | Negatieve staaf |
| S. mutants | Positieve kok |
| L. casei | Positieve staaf |
Biochemische Testen
Om verder te kunnen uitzoeken of de bacterie welke bacterie we gevonden hebben, zijn er biochemische technieken uitgevoerd. Te beginnen met de katalase. Hierin verwachten we dat het resultaat katalase negatief geeft voor de bacteriën waarnaar we opzoek zijn. Als er een positief resultaat gevonden wordt moet er een coagulase test worden uitgevoerd.
Coagulase
| Persoon | Witte kolonie | Groene kolonie |
|---|---|---|
| C | - | N.V.T. |
| F | N.V.T. | - |
Katalase
| Persoon | Witte kolonies | Groene kolonies |
|---|---|---|
| A | - | - |
| B | - | - |
| C | + | - |
| D | - | - |
| F | - | + |
| G | - | - |
| H | - | - |
API's
Ook zijn er API's uitgevoerd. Aan de hand van deze biochemische testen kan je op zoek gaan of je met je bacterie die je hebt wel de juiste in handen hebt waar je naar op zoek bent. Met de 20A zoek je specifiek de bacterie P. gingivalis en met de API 20 STREP de S. mutans. In de resultaten zie je dat de resultaten niet overeenkomen. Dit komt omdat de bacterie is overgezet van een bloedplaat. Op een bloedplaat kan van alles groeien. Het wordt ook wel een differentiële plaat genoemd. Dit heeft als nadeel dat wanneer je een bacterie wilt overzetten, je andere bacteriën mee kan nemen. De conclusie is dat de API's zijn mislukt doordat ze niet de juiste bacteriën geven en in de meeste gevallen geen 100% betrouwbaar resultaat geven.
| API 20A | Mogelijke bacterie/bacterien |
|---|---|
| D | Actinamyces naeslundii, Clastridium perifringens, Proplonibacterium proplonicum, Prexotella bivia |
| C | Bifidobacterium spp 2 |
| API 20 STREP | Mogelijke bacterie of bacteriën |
|---|---|
| B | Streptococcus uberis |
| G | Steptococcus uberis, Enterococcus faecium |
Nanodrop
Ter controle van de DNA isolatie wordt een nanodrop uitgevoerd. Het ratio wat wordt gegeven toont hoe zuiver het DNA is geïsoleerd. Een ratio van 1,8 is zuiver. De concentratie toon de hoeveelheid geïsoleerd materiaal. Hoe meer, hoe beter. Al heb je niks aan een hoge concentratie in combinatie met een laag ratio, omdat je dan alsnog geen zuiver DNA hebt. In de tabel valt op dat de concentraties heel laag zijn. Dit kan komen doordat er te weinig DNA is meegenomen voor de isolatie. Ook de ratio's liggen veelal onder de 1,8. Het DNA is dan niet zuiver geïsoleerd en nog vervuild met fenolen en eiwitten.
Uit de resultaten blijkt dus dat de ratio's niet allemaal bij de 1,8 zitten en dat ze dus vervuild zijn met eiwitten. Dit is natuurlijk wat minder omdat er makkelijker valt te werken met zuiver DNA.
| Persoon | Concentratie | A260/280 ratio |
|---|---|---|
| A | 17.7 | 1.49 |
| B | 12.6 | 1.24 |
| C | 13.1 | 1.46 |
| D | 9.4 | 1.53 |
| F | 4.3 | 1.89 |
| G | 7.7 | 1.53 |
| H | 8.3 | 1.52 |
Gelelektroforese na isolatie
Na de isolatie zijn de monsters op gel gezet te om te kijken of het DNA goed is geïsoleerd en dus een zichtbaar bandje of smeer moeten geven. Dit zou dan dus wit oplichtend te zien zijn tegen de zwarte achtergrond. Alle monsters waarbij er geen duidelijk bandje of smeer te zien is zijn hierbij afgevallen en worden daarom ook niet verder meegenomen in de rest van de experimenten. Deze zouden dus al te weinig DNA bevatten om mee verder te gaan.
Gelelektroforese na PCR met specifieke primers
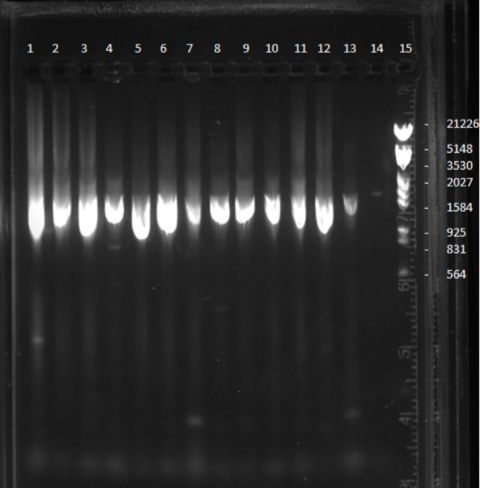
De monsters die wel oplichtten tijdens de gelelektroforese na de isolatie zijn meegenomen en geamplificeerd met een specifieke PCR. Aan de hand van specifieke primers kunnen de drie bacteriën worden aangetoond op de gel. Zo hebben we een primer voor de L. casei, P. gingivalis en de S.mutans gebruikt. Met deze primers zouden dus eigenlijk ook deze specifieke stukken DNA gevonden moeten worden van deze bacteriën. Aan de hand van een molmarker konden we de grootte van de bandjes bepalen. De molmarker dient dus eigenlijk als een legenda. Het bandje van de L. casei heeft een grote 118 bp, die van de S. mutans heeft een grote van 433 bp en die van P. gingivalis 404 bp.
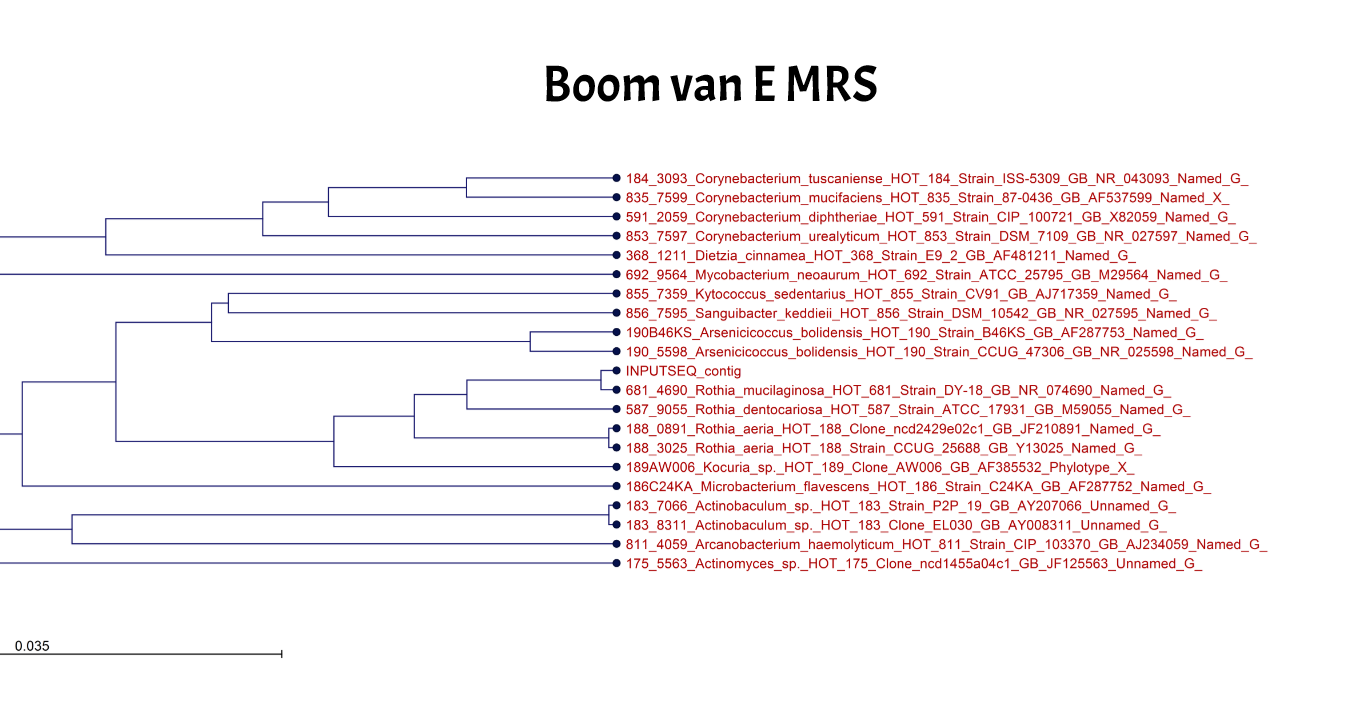
Deze gelelektroforese is na de PCR met specifieke primers uitgevoerd om te kijken of er ribosomaal DNA aanwezig is. Dit zit in bacteriën en op basis van dit DNA kan er onderscheid gemaakt worden tussen verschillende soorten. De resultaten zijn in de afbeelding te zien. Hierbij hebben we 12 monsters meegenomen en dit laat zien dat er bij alle 12 16s-rDNA aanwezig is. Hier zijn vier monsters van opgestuurd naar BaseClear om te laten sequensen. Dit sequensen houdt in dat de code van het DNA opgeschreven wordt zodat je precies weet wat er in het DNA zit. Deze uitkomst hebben we door een databank gehaald en hier zijn stambomen uit gekomen. Deze laten zien welke bacterie er precies gevonden is en hoeveel procent overeenkomst er is met de databank. Uit deze bomen blijkt dat testpersoon B 100% zeker Streptococcus salvarius heeft, persoon D had te weinig overeenkomsten tussen zijn DNA en de databank, E heeft 100% zeker de Rothia mucilaginosa en persoon F heeft 75% zeker Veillonella dispar.
Conclusie
Uit onze interventie is dus gebleken dat (bijna) alle proefpersonen een betere mondflora hebben ontwikkeld. Het maakt geen verschil tussen de groepen van Yakult light, het mondwater Listerine Zero of de controle groep. Dit komt mogelijk doordat de proefpersonen bewuster zijn gaan poetsen met de interventie in hun achterhoofd. We bevelen iedereen daarom ook aan om twee keer per dag te poetsen en eventueel nog te spoelen met een mondwater zonder alcohol.



Maak jouw eigen website met JouwWeb